
Vol. 36, n.º 3, 2003
REVISTA
ESPAÑOLA DE
Vol. 36, n.º 3, 2003 |
Ernesto Moro-Rodríguez1, M.ª Jesús Coma del Corral2, Miguel Sánchez-Ramos3
1 Universidad Rey Juan Carlos. Dpto. de Ciencias de la Salud. 2 Unidad de Investigación, Hospital General Yagüe. Burgos. 3 Hospital Materno Infantil. Las Palmas de Gran Canaria. jemoro@uninet.edu
RESUMEN
XML es un nuevo estándar de almacenamiento semiestructurado en el que, a diferencia del HTML, se pueden definir etiquetas o marcas personales por medio de una DTD (las Definiciones de Tipo de Documento o Restricciones de Domino) o mediante el uso de Schema. En los últimos años ha proliferado su uso como formato eficaz de almacenamiento e intercambio de datos dada su gran flexibilidad para poder representar diferentes clases de información. Para acceder a ésta información, extraerla y manipularla se han desarrollado también de forma paralela distintos lenguajes de consulta para XML como pueden ser: Xquery, Xpath, XQL, XML-QL y otros.
Responsabilidad última del patólogo es la producción y gestión de informes bajo los niveles adecuados de calidad, rapidez, fiabilidad y confidencialidad en base a adecuados medios de calidad e información. Para dar respuesta acertada a éstas obligaciones pensamos que en la actualidad existen soluciones entre las que se encontraría la de desarrollar un estándar de consenso en XML que pudiera servir para interconectar distintos gestores de bases de datos de patología con el que fueran posible, al menos, intercambiar un conjunto mínimo de datos al estilo del espíritu de los proyecto SIGNO pero desde la perspectiva actual que nos dan los tiempos.
Palabras clave: Patología, XML, estándares, bases de datos.
SUMMARY
XML is a new standard of semi-structured storing in which, unlike HTML, labels or personal marks can be defined through a DTD (Document Type Definition) or by means the use of Schema. Its use has spread over the last years as an efficient data storing and exchange format given its great flexibility to represent different classes of information. To access, extract and manipulate this data, different consultation languages for XML have been developed in parallel such as Xquery, Xpath, XQL, XML-QL and others.
The ultimate responsibility of pathologists is the production and management of reports with adequate quality, diligence, reliability and confidentiality levels through suitable resources for quality control and information management. To respond successfully to these obligations we think that solutions currently exist such as the development of a consensus XML standard useful for the interconnection of different data bases managers in the style of the inspiration of the SIGNO project but from an updated perspective.
Key words: Pathology, XML, standards, database.
¿Cuál es el interés o la necesidad que puede despertar a un patólogo acercarse a lo que se esconde bajo el acrónimo XML? Introducción y justificación
No es misión del presente trabajo definir lo que es XML («eXtensible Markup Language» o Lenguaje de Marca Extensible), cuanto más que va destinado a especialistas médicos dedicados al diagnóstico clínico-patológico cuyo área de conocimiento se escapa por completo de lo que pueda encerrarse bajo tal críptico acrónimo. El World Wide Web Consortium (W3C), que es la autoridad última en Internet encargada de ordenar los distintos estándares universalmente aceptados, documenta de forma precisa a través de su página oficial lo que ésta especificación encierra (1).
La pregunta formulada (¿Cuál es el interés o la necesidad que puede despertar a un patólogo acercarse a lo que se esconde bajo el acrónimo XML?) surge de la reflexión de lo que a diario supone la práctica profesional del patólogo, resumida por J. M. Sanz Anquela bajo el término «el producto del trabajo asistencial desarrollado en los Servicios de Anatomía Patológica son los diagnóstico realizados sobre especímenes de distinta procedencia y naturaleza...» (2). En el mismo Libro Blanco JF Martínez Tello desarrolló la «Cartera de Servicios Preliminares del Insalud» sobre la base de los Estudios Signo I y Signo II del Ministerio de Sanidad y Consumo enunciando que «el primer objetivo es el de disponer de un lenguaje común aplicado a un listado de todos los procedimientos diagnósticos que se pueden realizar en una Unidad Funcional Asistencia de Anatomía Patológica» y coincidía del mismo modo con Sanz-Anquela en que «desde el punto de vista asistencial, el patólogo realiza técnicas sobre especímenes para efectuar diagnósticos anatomopatológicos». Responsabilidad última del patólogo es por tanto la de la producción y gestión de éstos informes bajo los niveles adecuados de calidad, rapidez, fiabilidad y confidencialidad en base a adecuados medios de calidad e información (3).
Para llevar a cabo de forma correcta éstas responsabilidades, y dado el elevado volumen de información que debe producir y gestionar el patólogo, éste ha de requerir, en algún que otro momento, del uso de herramientas informáticas que le permitan registrar, organizar, actualizar, ordenar, buscar e imprimir esa información según sus necesidades y ajustándose además a las correspondientes normativas legales de cada momento.
En los pasados 25-30 años los distintos servicios de anatomía patológica de nuestro país han ido incorporando distintas tecnologías de bases de datos que, acordes a su tiempo, han podido resolver el problema de la creciente demanda de los estudios anatomopatológicos de nuestros hospitales.
Si bien las herramientas desarrolladas hasta la fecha han podido paliar gran parte de las exigencias que conlleva la responsabilidad del patólogo en cuanto a la producción y gestión de esa información; en el cotidiano quehacer de éstos profesionales queda todavía la insatisfacción de encontrarse ante un sistema imperfecto que no le aporta mucha más información que la que le refiera de forma puntual el clínico solicitante de los estudios de patología.
A esto se suma el hecho de que el paciente cada vez más hace uso de sus derechos en cuanto a la elección de la asistencia lo que puede ocasionar, en no raros casos, que las pruebas diagnósticas de patología se repitan innecesariamente en todos y cada uno de los centros hospitalarios por donde transita. Y lo que puede parecernos peor, con el desconocimiento de los profesionales que emiten dichos diagnósticos. Como consecuencias de ésta práctica surgen innumerables situaciones de compromiso, unas veces debidas a los distintos matices de interpretación a los que está sometido el propio diagnóstico morfológico, otras motivadas por la imposibilidad de poder contrastar en el momento preciso la totalidad de los diagnósticos emitidos, el contenido de sus informes o las preparaciones que les acompañan.
Si además observamos, a poco que nos hayamos movido por las distintas instituciones hospitalarias de nuestros país, que existe una dispar heterogeneidad de herramientas de gestión y protocolos de información aplicables a un mismo diagnóstico anatomopatológico, llegaremos a la conclusión de que, a pesar de que el ejercicio profesional del patólogo se basa en el reconocimiento preciso de cada una de las distintas entidades morfo-patológicas o procesos patológicos, el ansiado lenguaje común de la patología queda trastornado por un sinfín de matizaciones y «dialectos» que en algunos casos no permite entendernos adecuadamente.
Prueba de que esto es así resulta en la práctica el conocer que, por ejemplo, la migración de un sistema de gestión utilizado hasta una fecha dada a otro del que confiamos nos irá mejor, supone un esfuerzo técnico considerable cuando no quebraderos de cabeza en pensar si perderemos en un momento el trabajo almacenada durante tantos años.
En consecuencia, para dar solución a los problemas expuestos pensamos que en la actualidad existen soluciones que pueden, de uno u otro modo, contribuir a mejorar la situación descrita. Una de ellas sería el desarrollo de un estándar de consenso en XML que pudiera servir para interconectar distintos gestores de bases de datos de patología con el que fueran posible, al menos, intercambiar un conjunto mínimo de datos al estilo del espíritu de los proyecto SIGNO, pero desde la nueva perspectiva actual que los tiempos nos da.
Antecedentes históricos
En 1986 se definió el primer lenguaje de marca conocido como SGML (Standard Generalized Markup Language) si bien fue concebido con anterioridad entre los años 60 y 70 hasta que finalmente quedó estandarizado bajo la norma ISO 8879:1986. Seis años después la W3C publicó la primera especificación HTML como lenguaje destinado a ser usado para la creación de páginas web y doce años después se publicó la primera recomendación XML (4) actualizada en la versión 1.1 con fecha de 12 de Octubre de 2002 (5).
XML es un nuevo estándar de almacenamiento semiestructurado en el que, a diferencia del HTML, se pueden definir etiquetas o marcas personales por medio de una DTD (las Definiciones de Tipo de Documento o Restricciones de Domino) o mediante el uso de Schema. En los últimos años ha proliferado su uso como formato eficaz de almacenamiento e intercambio de datos dada su gran flexibilidad para poder representar diferentes clases de información. Para acceder a ésta información, extraerla y manipularla se han desarrollado también de forma paralela distintos lenguajes de consulta para XML como pueden ser: Xquery, Xpath, XQL, XML-QL y otros.
En la figura 1, procedente del documento http://www.w3.org/Consortium/, se nos muestra de forma esquemática las Recomendaciones propuestas por la W3 hasta la fecha, que alcanzan ya casi las 40, y en el que es sencillo observar cómo se está transformado la arquitectura inicial de la web (sustentada principalmente en el HTML, URLs y HTTPs) en una nueva arquitectura construida sobre el fundamento sólido aportado por XML.

Fig. 1: Esquema procedente de
http://www.w3.org/Consortium/, donde se nos muestra de forma esquemática las
Recomendaciones propuestas por la W3 hasta la fecha.
¿Qué propuestas similares a la formulada existen en la actualidad en Patología?
No resulta fácil encontrar una herramienta basada en XML que esté siendo usada de forma sistemática en Patología, salvo propuestas puntuales. Entre ellas cabría citar DocScope — Physician Friendly Medical Records software que está distribuido bajo Licencia Pública General (GPL) cuyo primer borrador fue liberado en mayo de 1998 por David Markwell (6). En la figura 2, procendente del la web de éste proyecto (http://openhealth.com/docscope/vision.html), se muestra el diagrama conceptual de la arquitectura propuesta por DocScope. En éste modelo el informe de cada paciente es conceptualmente un archivo en formato XML y cada archivo se trata como un objeto que puede ser manipulado mediante la API (Application Programming Interface) DOM (Document Object Model).
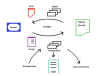
Fig. 2: Diagrama conceptual de la
arquitectura propuesta por DocScope.
Fanglin Zheng et al. en la 5.ª Conferencia Anual APIII (Advancing Pathology Informatics, Imaging, and the Internet) presentaron la propuesta PARMeta (Pathology Report Metadata) como un Modelo de Metadatos Inicial y su DTD/XML para informes en Patología Quirúrgica. PARMeta incluye no una única y monolítica DTD, sino una DTD principal y una librería de DTDs secundarias o «ramas» (las cuales podrían tener sus propias ramas). Los elementos de datos básicos de la DTD principal derivan desde el UPMC Copath C/S hasta el MARS HL7 en su versión 2.2. La estructura principal incluye elementos bastante constantes como pueden ser, por ejemplo, el número de biopsia o el diagnóstico pre-operatorio. Las DTD ramas las definen usando tres parámetros: enfermedad (p.e. maligno versus benigno), órgano (p.e. próstata versus otros) y procedimiento (p.e. resección versus biopsia versus TURP) e incluye los elementos de datos específicos a una enfermedad dada, órgano y procedimiento. Tanto un ser un ser humano como una máquina podrían construir una DTD completa y válida para cualquier objetivo deseado mediante la selección apropiada de las DTDs secundarias (7).
Ralf Schweiger et al en el Congreso XML 2000 Europe desarrollaron una aproximación flexible en XML basada sobre un concepto de plantilla y que permitía a la ingeniería de las aplicaciones adaptarse de forma rápida a la adquisición de la información (la data) y al almacenamiento estructurado dentro de las necesidades clínica cambiantes (8).
Steve Pynegar en el Congreso XML 2001 Europe presentó su experiencia relacionada con el denominado XML Pathology Requesting Project que está en uso actualmente en Doncaster Royal Infirmary (9) basado sobre el estándar inglés EDIFACT (10).
«The Open Healthcare Group» es una organización dedicada a la promoción y a la distribución de XChart, un programa de código abierto útil en el campo de la salud que está basado en tecnología XML. A través de la página web de éste proyecto se puede acceder a varias demos de ésta aplicación (http://www.openhealth.org/opnote/) entre las que cuentan con un sistema de registros de informes de biopsias del SNC obtenidas mediante esterotaxis (fig. 3) (11). Las DTDs propuestas por ésta organización se encuentran en la URL: http://www.openhealth.org/ASTM/overview.htm entre las que ésta la pathology.report.dtd destinada a nuestra disciplina.
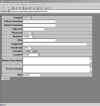
Fig. 3: The Open Healthcare Group. Sistema
de registros mediante Xchart para informes de biopsias del SNC obtenidas
mediante esterotaxis.
En la literatura científica son escasas también las referencias que pueden encontrarse sobre éste tema, entre ellas valdría citar las experiencias comunicadas independientemente por Akiyaman en Japón (12) y Brelstaff en Italia (13).
Finalmente referir que desde la web de ASTM International se pueden encontrar varios documentos y propuestas como E2182-02 Standard Specification for Clinical XML DTDs in Healthcare que pueden servir de ayuda a la hora de adaptar distintas DTDs en el campo de la salud (14).
Un ejemplo práctico de base de datos para gestión de informes de patología mamaria con implementación XML
Algunos gestores de bases de datos comerciales incorporan ya la posibilidad de publicar bases de datos en la World Wide Web o en una Intranet. Este es el caso de FileMaker Pro (por ejemplo, que gracias a un plug-in, Web Companion, sin la necesidad de usar otro software adicional, permite visualizar, editar, borrar o buscar en una base de datos por medio de cualquier navegador y siempre que se disponga de los privilegios adecuados de acceso. Además, también va siendo cada vez más frecuente que éste tipo de gestores puedan intercambiar la información de sus registros mediante XML o visualizarla mediante hojas de estilo XSLT. Siguiendo éste procedimiento hemos creado un ejemplo práctico de base de datos para gestionar informes de patología mamaria basado en el sistema de informe tipo del AFIP sobre la histopatología de la mama (15). Se puede acceder a ésta base de datos de prueba desde la URL: http://eusalud.uninet.edu/PTHML/examples.html.
En las figuras 4 y 5 podemos observar la forma como se visualizan los registros en la base de datos y los distintos menús y controles para poder añadir nuevos registros o introducir y cambiar los datos en cada uno de los campos. En la figura 6 se muestra como se puede acceder a la base de datos a través de un navegador web mientras que la figura 7 podemos observar parte del código exportado por la base de datos en formato FMPXML.

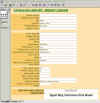
Figs. 4 y 5: Forma de como se visualizan
los registros en la base de datos ejemplo y los distintos menús y controles
para poder añadir nuevos registros o introducir y cambiar los datos en cada uno
de los campos.
Sirva el presente ejemplo de muestra para darnos cuenta de que ésta tecnología puede estar al alcance de todos, sin necesidad de descender, si no se quiere, al nivel de desarrollar una DTD específica para nuestras necesidades (aunque pensamos que éste sería el objetivo final si quisiéramos estandarizar nuestro lenguaje). Cualquier informe tipo es subsidiario de poder ser tabulado o concretado en una estructura de base de datos por campos. Tarea al margen y ya propia de los especialistas en anatomía patológica sería la de seleccionar aquellos informes tipo que mejor se adecuen a la evaluación de los distintos procesos patológicos; pero en sí, el sistema de creación, diseño y gestión de las bases de datos sigue un proceso perfectamente adaptable a las necesidades que se precisen en cada momento. La principal ventaja que una implementación XML puede añadir a estos sistemas será la posibilidad de exportar los registros en un estándar abierto y fácilmente intercambiable con otras bases de datos propietarias o de software libre.
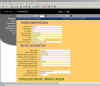
Fig. 6: Se muestra como puede accederse a
la base de datos ejemplo a través de un navegador web.
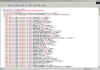
Fig. 7: Podemos observar parte del código
exportado por la base de datos ejemplo en formato FMPXML.
¿Cómo poder articular en nuestro medio una iniciativa de éstas características?
Conocemos por experiencia que la formulación de una empresa de éstas características en nuestro medio no resulta fácil. Por un lado, los licenciatarios propietarios de software en nuestro país no son ajenos a éste tipo de solicitudes y de forma unilateral pretenden dar respuesta a lo demandado para su aplicación concreta. Sin embargo, sigue sin haber un tutor que rija las normas de su legítimo juego.
Entendemos que los rectores que deberían enarbolar la batuta para orquestar el actual desconcierto serían distintos grupos de trabajo dentro de nuestras sociedades científicas que, en contacto con los distintos colectivos de profesionales implicados e interesados por éste tema, hicieran ver a las empresas licenciatarias del software la necesidad de abordar propuestas como las aquí expuestas.
Sería por tanto deseable que desde el seno de nuestras organizaciones profesional y sociedades científicas se aglutinara de alguna forma el esfuerzo encaminado a ponernos de acuerdo en proponer, a quien corresponda tomar una decisión de mayor alcance, al menos, ese conjunto mínimo de información que pudiera ser intercambiado entre distintas bases de datos bajo una misma Definición de Tipo de Documento (DTD); al tiempo que requerir a nuestros licenciadores de software que respetaran esa especificación y si lo desean colaborar en alcanzar ese consenso.
Con el ánimo de contribuir a fomentar ese diálogo ponemos a disposición de todo aquel que lo desee la lista de distribución XML-PTHML gestionada desde Uninet en la URL: http://listas.uninet.edu/mailman/listinfo/pthml donde pueden suscribirse y compartir los comentarios que haya suscitado la lectura del presente documento.
BIBLIOGRAFÍA
Extensible Markup Language (XML) http://www.w3.org/XML/
Sanz Anquela JM et al. Evaluación de la presión asistencia en los servicios de Anatomía Patológica de 44 Hospitales Españoles. Libro Blanco de la Anatomía Patológica en España. (Primer suplemento). Sociedad Española de Anatomía Patológica. 1999.
Martínez Tello FJ. Objetivos de la anatomía patológica. Cartera de Servicios. Libro Blanco de la Anatomía Patológica en España. (Primer suplemento). Sociedad Española de Anatomía Patológica. 1999.
Extensible Markup Language (XML) 1.0 W3C Recommendation 10-February-1998 http://www.w3.org/TR/1998/REC-xml-19980210
Extensible Markup Language (XML) 1.1 W3C Candidate Recommendation 15 October 2002 http://www.w3.org/TR/xml11/
DocScope — Physician Friendly Medical Records. http://openhealth.com/docscope/
Fanglin Zheng. An Initial Metadata Model and Its DTD/XML for Surgical Pathology Report. The 5th Annual Conference Advancing Pathology Informatics, Imaging, and the Internet. http://apiii.upmc.edu/apiii2000/ab-pitt3.htm
Schweiger R, Tafazzoli A. Using XML for flexible data entry in healthcare. Example use for pathology. XML 2000 Europe Conference Proceedings. http://www.infoloom.com/gcaconfs/WEB/paris2000/S32-05.HTM#N19
Pynegar S: XML and Pathology. XML 2001 Europe Conference Proceedings. http://www.gca.org/papers/xmleurope2001/papers/html/sid-04-09.html
NHA Information Authority. Implementing National Standars Pathology Reports Messagin http://www.nhsia.nhs.uk/pathology/pages/edifact.asp?om=m6
The Open Healthcare Group. http://www.openhealth.org/index.htm
Akiyama M. Migration of the Japanese healthcare enterprise from a financial to integrated management: strategy and architecture. Medinfo. 2001; 10: 715-718.
Brelstaff G, Moehrs S, Anedda P, Tuveri M, Zanetti G. Internet patient records: new techniques. J Med Internet Res. 2001; 3: E8.
E2182-02 Standard Specification for Clinical XML DTDs in Healthcare. http://www.astm.org/cgi-bin/SoftCart.exe/DATABASE.CART/REDLINE_PAGES/E2182.htm?L+mystore+wnch1655+1052439705
Fattaneh A. Tavassoli. Specimen Processing in Pathology of the Breast. Second Ed. 1999, Appleton & Lange. Stamford, Connecticut. Pp 110.